# Pytorch-Medical-Segmentation(2D 3D)
**Repository Path**: fxtfxt/Pytorch-Medical-Segmentation
## Basic Information
- **Project Name**: Pytorch-Medical-Segmentation(2D 3D)
- **Description**: No description available
- **Primary Language**: Unknown
- **License**: MIT
- **Default Branch**: master
- **Homepage**: None
- **GVP Project**: No
## Statistics
- **Stars**: 3
- **Forks**: 1
- **Created**: 2021-06-01
- **Last Updated**: 2024-02-22
## Categories & Tags
**Categories**: Uncategorized
**Tags**: None
## README
# Pytorch Medical Segmentation
英文版请戳:这里!
## 最近的更新
* 2021.1.8 训练和测试代码已经发布
* 2021.2.6 修复计算dice的一个bug,感谢[Shanshan Li](https://github.com/ssli23)的帮助~
* 2021.2.24 发布一个视频教程(https://www.bilibili.com/video/BV1gp4y1H7kq/)。
* 2021.5.16 修复Unet3D实现的错误。
* 2021.5.16 评估代码公布。
## 环境要求
* pytorch1.7
* torchio<=0.18.20
* python>=3.6
## 通知
* 您可以修改**hparam.py**文件来确定是2D分割还是3D分割以及是否可以进行多分类。
* 我们几乎提供了所有的2D和3D分割的算法。
* 本项目兼容几乎所有的医学数据格式(例如 nii.gz, nii, mhd, nrrd, ...),修改**hparam.py**的**fold_arch**即可。
* 如果您想进行**多分类**分割,请自行修改下列代码。我不能确定您的具体分类数。
* https://github.com/MontaEllis/Pytorch-Medical-Segmentation/blob/48edef7751af8551b7432b5491f4cf1964bd0cfc/hparam.py#L6
* https://github.com/MontaEllis/Pytorch-Medical-Segmentation/blob/48edef7751af8551b7432b5491f4cf1964bd0cfc/main.py#L235
* https://github.com/MontaEllis/Pytorch-Medical-Segmentation/blob/48edef7751af8551b7432b5491f4cf1964bd0cfc/main.py#L336
* https://github.com/MontaEllis/Pytorch-Medical-Segmentation/blob/48edef7751af8551b7432b5491f4cf1964bd0cfc/main.py#L496
* https://github.com/MontaEllis/Pytorch-Medical-Segmentation/blob/48edef7751af8551b7432b5491f4cf1964bd0cfc/data_function.py#L69
* https://github.com/MontaEllis/Pytorch-Medical-Segmentation/blob/48edef7751af8551b7432b5491f4cf1964bd0cfc/data_function.py#L167
* 不论是2D或是3D,本项目均采用**patch**的方式。故图片大小不必严格保持一致。
## 准备您的数据
### 例1
如果您的source文件夹如下排列 :
```
source_dataset
├── source_1.mhd
├── source_1.zraw
├── source_2.mhd
├── source_2.zraw
├── source_3.mhd
├── source_3.zraw
├── source_4.mhd
├── source_4.zraw
└── ...
```
同时您的label文件夹如下排列 :
```
label_dataset
├── label_1.mhd
├── label_1.zraw
├── label_2.mhd
├── label_2.zraw
├── label_3.mhd
├── label_3.zraw
├── label_4.mhd
├── label_4.zraw
└── ...
```
您应该修改 **fold_arch** 为 **\*.mhd**, **source_train_dir** 为 **source_dataset** 并修改 **label_train_dir** 为 **label_dataset** in **hparam.py**
### Example2
如果您的source文件夹如下排列 :
```
source_dataset
├── 1
├── source_1.mhd
├── source_1.zraw
├── 2
├── source_2.mhd
├── source_2.zraw
├── 3
├── source_3.mhd
├── source_3.zraw
├── 4
├── source_4.mhd
├── source_4.zraw
└── ...
```
同时您的label文件夹如下排列 :
```
label_dataset
├── 1
├── label_1.mhd
├── label_1.zraw
├── 2
├── label_2.mhd
├── label_2.zraw
├── 3
├── label_3.mhd
├── label_3.zraw
├── 4
├── label_4.mhd
├── label_4.zraw
└── ...
```
您应该修改 **fold_arch** 为 **\*/\*.mhd**, **source_train_dir** 为 **source_dataset** 并修改 **label_train_dir** 为 **label_dataset** in **hparam.py**
## 训练
* 不使用预训练模型
```
set hparam.train_or_test to 'train'
python main.py
```
* 使用预训练模型
```
set hparam.train_or_test to 'train'
python main.py -k True
```
## Inference
* 测试
```
set hparam.train_or_test to 'test'
python main.py
```
## 实例
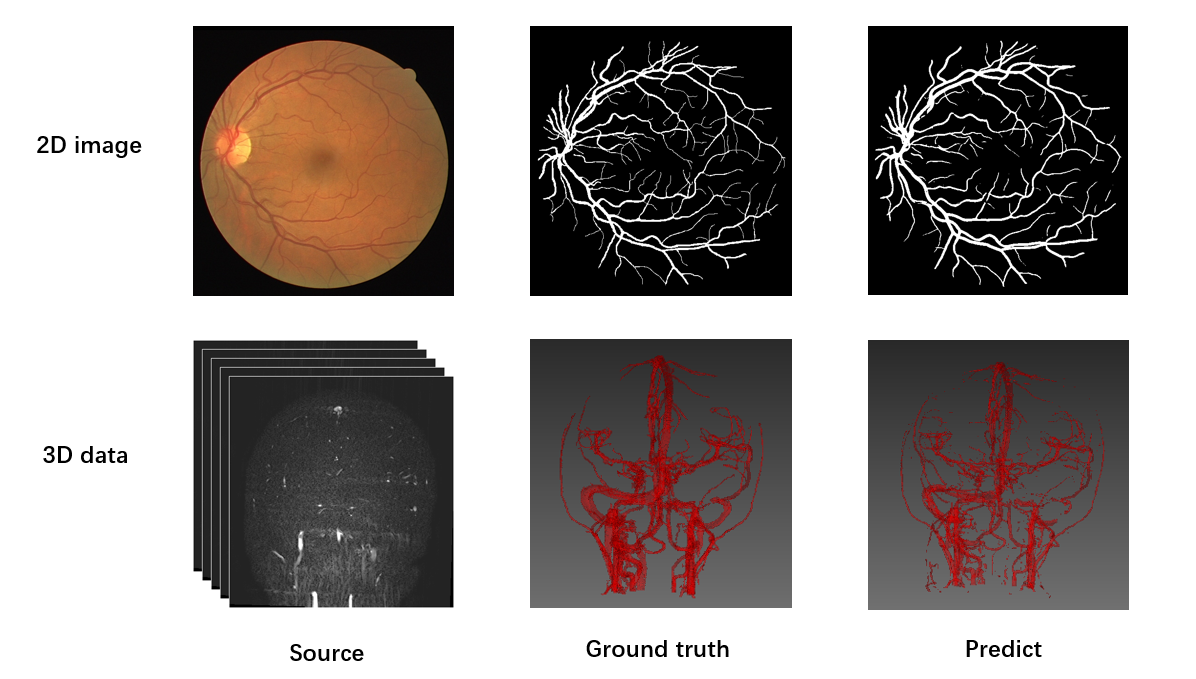
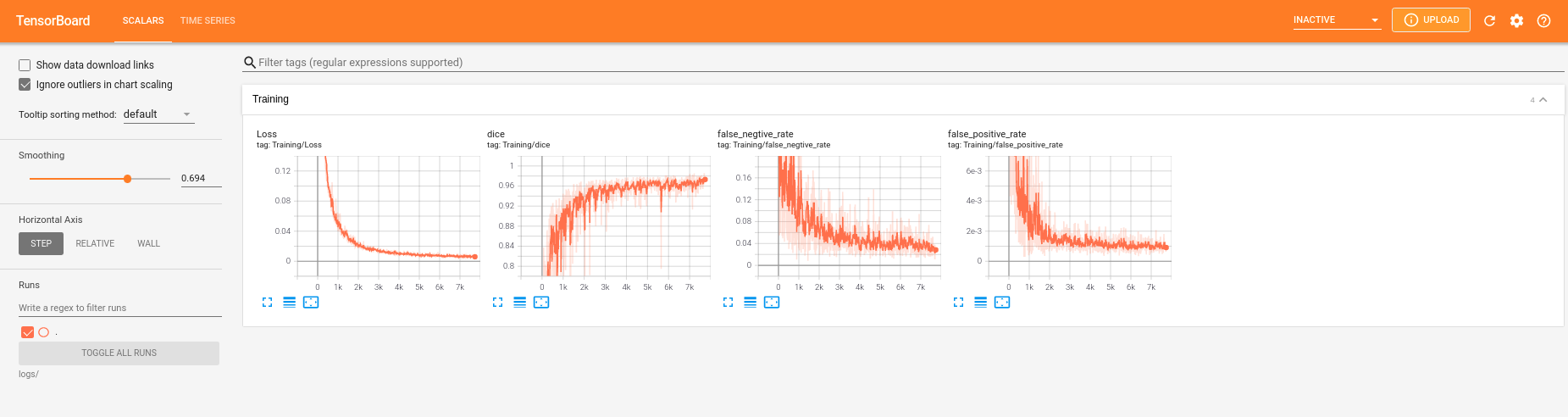
## 教程
* https://www.bilibili.com/video/BV1gp4y1H7kq/
## Done
### Network
* 2D
- [x] unet
- [x] unet++
- [x] miniseg
- [x] segnet
- [x] pspnet
- [x] highresnet(copy from https://github.com/fepegar/highresnet, Thank you to [fepegar](https://github.com/fepegar) for your generosity!)
- [x] deeplab
- [x] fcn
* 3D
- [x] unet3d
- [x] residual-unet3d
- [x] densevoxelnet3d
- [x] fcn3d
- [x] vnet3d
- [x] highresnert(copy from https://github.com/fepegar/highresnet, Thank you to [fepegar](https://github.com/fepegar) for your generosity!)
- [x] densenet3d
### Metric
- [x] metrics.py 来评估您的结果
## TODO
- [ ] dataset
- [ ] benchmark
- [ ] nnunet
## By The Way
这个项目并不完美,还存在很多问题。如果您正在使用这个项目,并想给作者一些反馈,您可以给[Kangneng Zhou](elliszkn@163.com)发邮件,或添加他的**微信**:ellisgege666
## 致谢
这个项目是一个非官方PyTorch实现的3D和2D医学分割,高度依赖于[MedicalZooPytorch](https://github.com/black0017/MedicalZooPytorch)和[torchio](https://github.com/fepegar/torchio)。感谢上述项目。感谢[Cheng Chen](b20170310@xs.ustb.edu.cn), [Daiheng Gao](https://github.com/tomguluson92), [Jie Zhang](jpeter.zhang@connect.polyu.hk), [Xing Tao](kakatao@foxmail.com), [Weili Jiang](1379252229@qq.com)和[Shanshan Li](https://github.com/ssli23) 对我的帮助。